Am Zentrum für Biotechnologie (CeBiTec) der Universität Bielefeld wird eine komplette Cloud-Lösung für Bioinformatiker zur Verfügung stehen. Sie umfasst eine Rechen- und Speicherinfrastruktur (IaaS), ein Framework für den vereinfachten Zugang zur Cloud-Infrastruktur (PaaS) und gebrauchsfertige bioinformatische Workflow-Lösungen (SaaS) im Bereich der Metagenomik. Die bereitgestellte Cloud-Umgebung umfasst die folgenden Dienste:
- Infrastructure as a Service (IaaS): Sehr fortgeschrittene Benutzer beherrschen die Tools zur Erstellung von Workflows und Pipelines, die eine leistungsstarke Infrastruktur für die Datenanalyse erfordern. Die Benutzer greifen über das Virtualisierungssystem auf die Infrastruktur zu, z. B. über virtuelle Maschinen oder Docker-Container.
- Plattform-as-a-Service (PaaS): Wir bieten BiBiGrid als Tool für die einfache Einrichtung von HPC-Clustern in einer Cloud-Umgebung an. Es vereinfacht die oft komplexe Konfiguration und Einrichtung typischer Software-Stacks auf einen einzigen Befehlszeilenaufruf.
- Software as a Service (SaaS): Ein Hauptziel des de.NBI-Cloud-Standorts in Bielefeld ist es, beliebte Bioinformatik-Workflows für Cloud-Benutzer verfügbar zu machen.
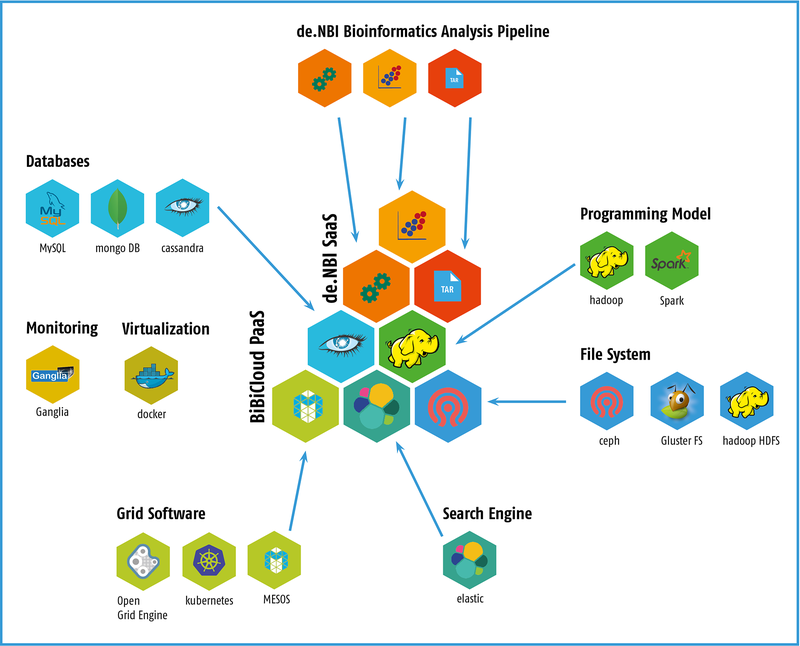
Beliebte Bioinformatik-Tools und Pipelines werden über vorkonfigurierte VMs oder Docker-Container zur Verfügung gestellt. Der Schwerpunkt des Bielefelder Service Centers liegt auf mikrobieller Genomik und Metagenomik sowie auf Postgenomik-Anwendungen. Innerhalb der de.NBI-Cloud werden Datensätze und vorkonfigurierte Pipelines für Analysen in diesen Forschungsbereichen einem breiten Publikum zur Verfügung gestellt. Dazu gehören im Bereich der Metagenomik sowohl 16S rRNA-basierte als auch WGS-basierte Analyse-Workflows, einschließlich groß angelegter Assemblierungen und funktioneller Annotation. Für diese speziellen Anwendungen können VMs mit bis zu 2 TB RAM bereitgestellt werden.
Ein besonderer Schwerpunkt ist auch die Analyse von Oxford Nanopore Sequenzdaten und Postgenomics-Anwendungen, wo wir z.B. Ressourcen für den MetaProteomeAnalyzer Service (MetaProtServ) bereitstellen.
Die de.NBI Cloud-Infrastruktur an der Universität Bielefeld umfasst neben allgemeinen Ressourcen auch Instanzen mit hohem Arbeitsspeicher und lokalem Festplattenspeicher (bis zu 2 TB RAM und 5 TB lokaler Festplattenspeicher) für Anwendungen wie metagenomische Assemblierungen. Für hardwarebeschleunigte Anwendungen des maschinellen Lernens sind spezielle VMs mit Zugriff auf GPUs verfügbar.