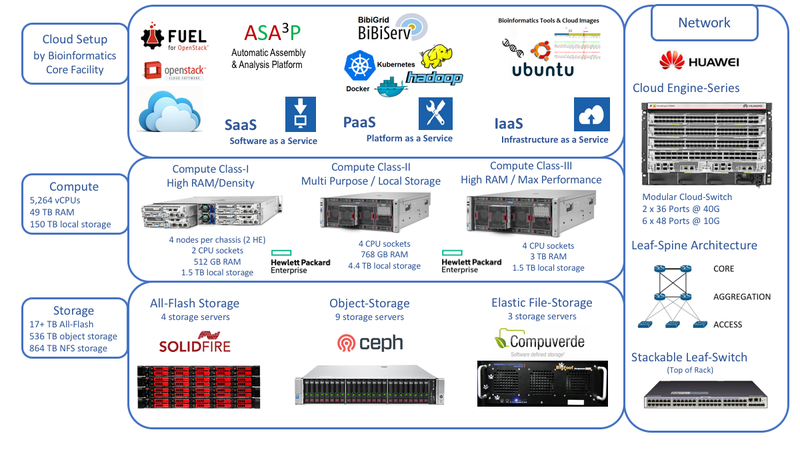
Die de.NBI-Cloud-Infrastruktur an der Justus-Liebig-Universität (JLU) Gießen wurde Anfang 2017 erfolgreich eingerichtet und stellt den Nutzern des de.NBI-Netzwerks umfangreiche Rechen- und Speicherressourcen zur Verfügung. Eine Erweiterung der gesamten Speicherkapazität ist bereits im Gange und wird in der ersten Jahreshälfte 2018 verfügbar sein.
Nach der Ersteinrichtung und intensiven Tests wurde die Cloud an der JLU Gießen im Juni 2017 in den Produktionsmodus überführt. Sie wurde bereits erfolgreich für die Durchführung praktischer Sitzungen während der ersten de.NBI-Sommerschule zum Thema Cloud Computing für die Bioinformatik genutzt. Bei dieser Gelegenheit wandten 24 Teilnehmer Cloud-Computing-Techniken für die Analyse ihrer eigenen biologischen Datensätze an und lernten, wie man skalierbare Bioinformatik-Softwarelösungen in einer Cloud-Umgebung implementiert. Seit ihrer Einrichtung wird die Cloud regelmäßig von verschiedenen Kooperationspartnern und Nutzern des de.NBI-Netzwerks genutzt, um ihre Daten zu analysieren.
Mit zwischen 6 und 27 GB Hauptspeicher pro CPU-Kern bietet die großzügige Ausstattung der de.NBI-Cloud an der JLU Gießen eine ideale Ressource für die Ausführung von Tools und Workflows mit hohem Speicherbedarf, wie z.B. die Assemblierung großer Metagenome oder eukaryotischer Genome. Zu diesem Zweck werden Instanztypen mit bis zu 3 TB Hauptspeicher und über 100 CPU-Kernen angeboten.
Die Rechenressourcen für die Ausführung virtueller Maschinen werden durch eine Auswahl verschiedener Speicherlösungen ergänzt, darunter SSD-basierte ephemere Festplatten, Datenvolumina und Objektspeicher, die zur Speicherung von Roh- und Zwischendaten sowie der Endergebnisse von bioinformatischen Analysepipelines verwendet werden können.
Das de.NBI-Cloud-Team an der JLU Gießen bietet gebrauchsfertige Bilder für die Analyse, Assemblierung und statistische Verarbeitung mikrobieller Daten. Außerdem werden wir in Kürze verschiedene vorselektierte und sofort nutzbare biologische Datenbanken und Sequenzsammlungen wie NCBI GenBank, RefSeq, Pfam oder vorverarbeitete menschliche Genome anbieten, die über ein gemeinsames Dateisystem zur sofortigen Nutzung zur Verfügung gestellt werden. Auf diese Weise können die Benutzer sofort mit der Analyse ihrer Daten beginnen, ohne dass sie die erforderlichen Datenbanken mühsam zusammensuchen und vorverarbeiten müssen.
In Zusammenarbeit mit unseren BiGi-Partnern an der Universität Bielefeld steht das BiBiGrid-Framework zur Verfügung, das eine einfache Möglichkeit bietet, Cloud-basierte Computing-Cluster einzurichten, um große Datenmengen parallel zu verarbeiten, wie sie z.B. für die Verarbeitung von Metagenom- oder Transkriptomdaten erforderlich sind. Zusätzlich zum BiBiGrid-Framework bieten wir eine der jüngsten Ergänzungen unseres Portfolios an Bioinformatik-Anwendungen an, die ASAP-Anwendung, die für die gleichzeitige Analyse, Assemblierung, Annotation und vergleichende Analyse von bakteriellen Genomen verwendet werden kann. Auf der Grundlage dieser einzigartigen Kombination und mit Hilfe der de.NBI-Cloud an der JLU Gießen sind wir derzeit in der Lage, problemlos Tausende von mikrobiellen Genomen pro Tag zu analysieren.
Zusätzlich zu den Allzweck-Computing-Servern verfügen wir über mehrere ActiveMotif Decypher FPGA-basierte Systeme, die hardwarebeschleunigte Versionen ausgewählter Bioinformatik-Anwendungen bereitstellen, z. B. Sequenzhomologiesuchen, die für die Genomanalyse und -annotation sowie die Verarbeitung von Metagenomdaten erforderlich sind. Derzeit evaluieren wir verschiedene Lösungen, um diese Systeme auch unseren Cloud-Nutzern zur Verfügung zu stellen. Darüber hinaus werden wir in der ersten Jahreshälfte 2018 eine vereinfachte Bereitstellung von elastischen und flexiblen Datenanalyseumgebungen auf Hadoop-Basis anbieten, die von jedem einzelnen Nutzer angepasst werden können.
Im Sommer 2017 umfasste das de.NBI-Cloud-Setup in Gießen etwa 80 Hosts mit mehr als 2.600 Kernen, insgesamt 48 TB RAM, 140 TB lokalen SSD-Speicher in den Compute-Hosts und etwa 500 TB verteilten Speicher für Volumes und Objektspeicher.